大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
近日,英国剑桥大学Courtney W. Hanna团队和西班牙马德里自治大学Vicente Perez-Garcia团队合作,在Cell Stem Cell杂志发表题为《Ectopic expression of DNMT3L in human trophoblast stem cells restores features of the placental methylome》的研究论文,研究通过微量全基因组重亚硫酸盐测序(WGBS)、染色质免疫共沉淀测序(ChIP-seq)、转录组测序(RNA-seq)等揭示了人滋养层干细胞(hTSCs)的DNA甲基化及其在胎盘发育中的作用,并证明DNMT3L能够恢复hTSCs中胎盘的部分甲基化区域(PMDs)和调控滋养层细胞分化,为胎盘功能及相关妊娠疾病的研究提供了新依据。

标题:Ectopic expression of DNMT3L in human trophoblast stem cells restores features of the placental methylome(人滋养层干细胞中DNMT3L异位表达可恢复胎盘甲基化表征)
发表时间:2025年2月6日
发表期刊:Cell Stem Cell
影响因子:IF20.4/Q1
技术平台:微量WGBS、ChIP-seq、RNA-seq等(易基因金牌技术)
作者单位:英国剑桥大学、西班牙马德里自治大学
DOI:10.1016/j.stem.2024.12.007
胎盘的DNA甲基化图谱具有特异性且广泛存在部分甲基化区域(PMDs)。胎盘“甲基化组”在哺乳动物中高度保守,是许多癌症鉴定的共有表征,与妊娠并发症的关联受到广泛研究。人类滋养层干细胞(hTSCs)为胎盘功能研究提供了巨大潜力,有助于更好理解表观遗传特征;然而hTSCs表观基因组是否能够模拟原代滋养层细胞不完全清楚。本研究发现,与滋养层外细胞(Trophectoderm,TE)和第一孕期(孕早期,孕1-12周)滋养层细胞(Cytotrophoblast,CTBs)相比,hTSCs表现出不典型的甲基化组。无论细胞来源、氧气水平还是培养条件如何,hTSCs均显示出在转录基因体内的局部DNA甲基化,并且完全失去PMDs。与早期人类滋养层细胞不同,hTSCs的DNMT3L表达显著缺失,而小鼠滋养层细胞中PMDs的建立依赖于DNMT3L。研究证明在hTSCs中异位表达DNMT3L可以恢复胎盘PMDs的DNA甲基化,支持DNMT3L在人类早期胚胎发生过程中滋养层发育的de novo甲基化保守。
- hTSCs与第一孕期滋养层细胞(1st CTBs)相比,在胎盘PMDs区域表现出低甲基化。
- 细胞来源和培养条件对hTSCs的DNA甲基化模式作用有限。
- DNMT3L在人类滋养层细胞的de novo甲基化可能窗口期间表达。
- 在hTSCs中异位表达DNMT3L可恢复胎盘PMDs的DNA甲基化。
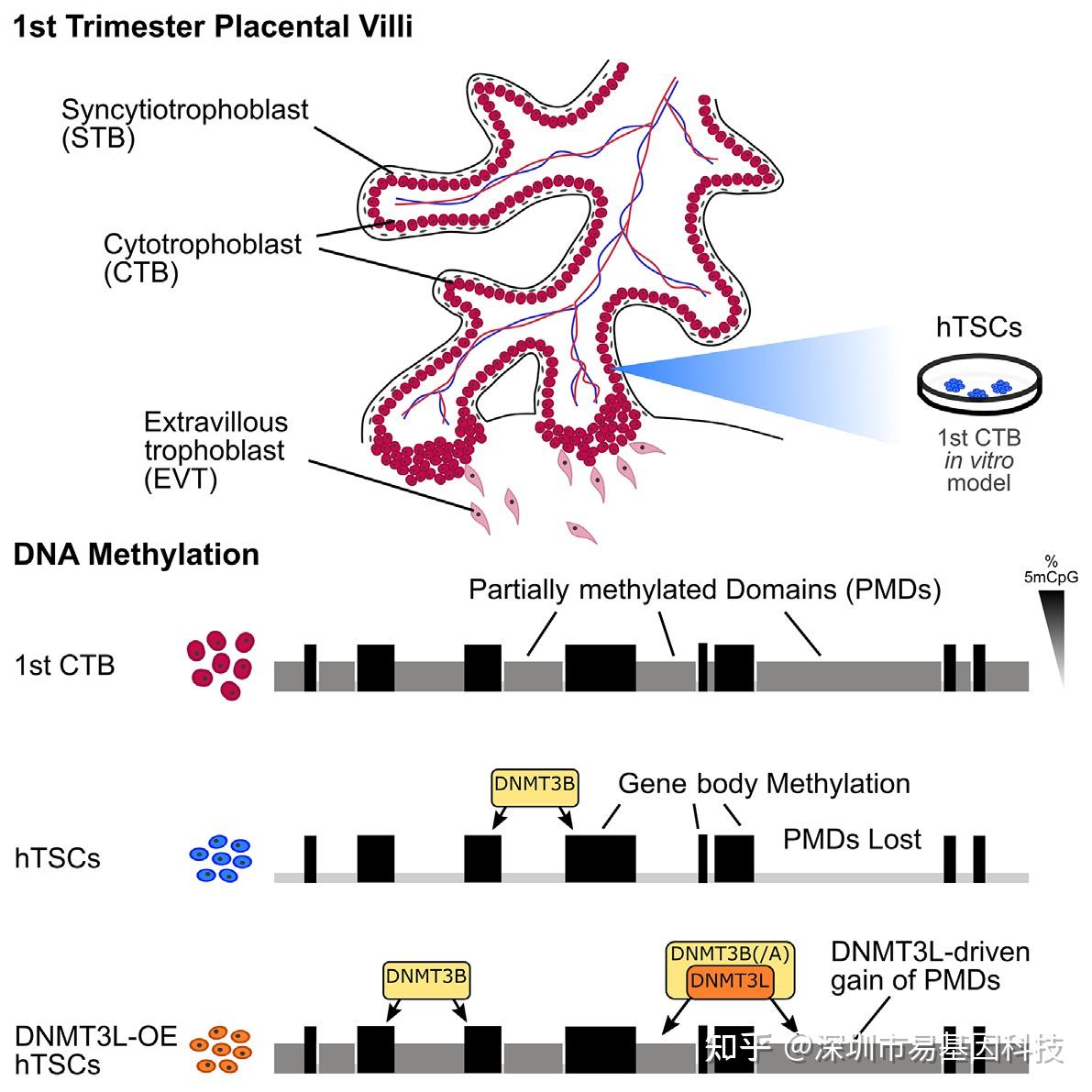
图形摘要
易基因相关拓展性产品案例
- Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
- 项目文章 | 植入前胚胎的全基因组DNA甲基化和转录组分析揭示水牛胚胎基因组激活进展
- 项目文章|微量WGBS+ACE-seq揭示卵巢早衰的人卵丘细胞DNA甲基化与羟甲基化表观基因组图谱
- 项目文章 | JGG:单细胞甲基化+转录组多组学分析揭示哺乳期母体低蛋白饮食对子代的跨代传递
- 项目文章:微量DNA甲基化分析揭示MeCP2在卵子发生和卵巢衰老中的表观遗传调控
- Nature | 易基因DNA甲基化测序助力人多能干细胞向胚胎全能8细胞的人工诱导
研究方法
样本收集与处理:从6日龄人类囊胚中收集滋养层外细胞(TE),并从第一孕期胎盘中分离出滋养层细胞(CTBs),并培养囊胚(BTS11)和孕早期CTB(CT29)的hTSCs。
DNA甲基化分析:利用微量WGBS技术,分析TE、CTBs和hTSCs的全基因组DNA甲基化模式。将这些细胞类型的DNA甲基化图谱与已发表的数据集进行比较,揭示hTSCs与原代滋养层细胞之间的差异。
细胞培养与分化:研究了不同培养条件(如氧气浓度、培养基类型)对hTSCs DNA甲基化模式的影响。诱导hTSCs分化为合体滋养层细胞和细胞滋养层细胞,评估DNA甲基化模式变化。
DNMT3L过表达实验:通过慢病毒介导的DNMT3L过表达,研究DNMT3L对hTSCs DNA甲基化模式的影响,特别是对胎盘PMDs的恢复作用。并评估DNMT3L过表达hTSCs在类器官培养中的分化能力。
ChIP-seq:分析hTSCs中的组蛋白修饰,特别是H3K4me3、H3K36me3和H3K27me3的分布。
基因表达分析:通过RNA-seq和RT-qPCR技术检测hTSCs中的基因表达水平。
结果图形
(1)人滋养层干细胞(hTSCs)表现出与滋养外胚层细胞(TE)和CTBs的差异DNA甲基化特征表征
研究发现,hTSCs的DNA甲基化模式与TE和CTB存在显著差异。与TE和CTB相比,hTSCs显示出较低的DNA甲基化水平,且胎盘特有的部分甲基化区域(PMDs)缺失,这些PMDs在CTBs中表现为部分甲基化状态(20%-60%)。此外,hTSCs的DNA甲基化主要集中在转录基因体内,与基因表达水平呈正相关(Figure 1)。这一结果表明,hTSCs的DNA甲基化模式与原代滋养层细胞存在显著差异,可能影响其功能和分化能力。
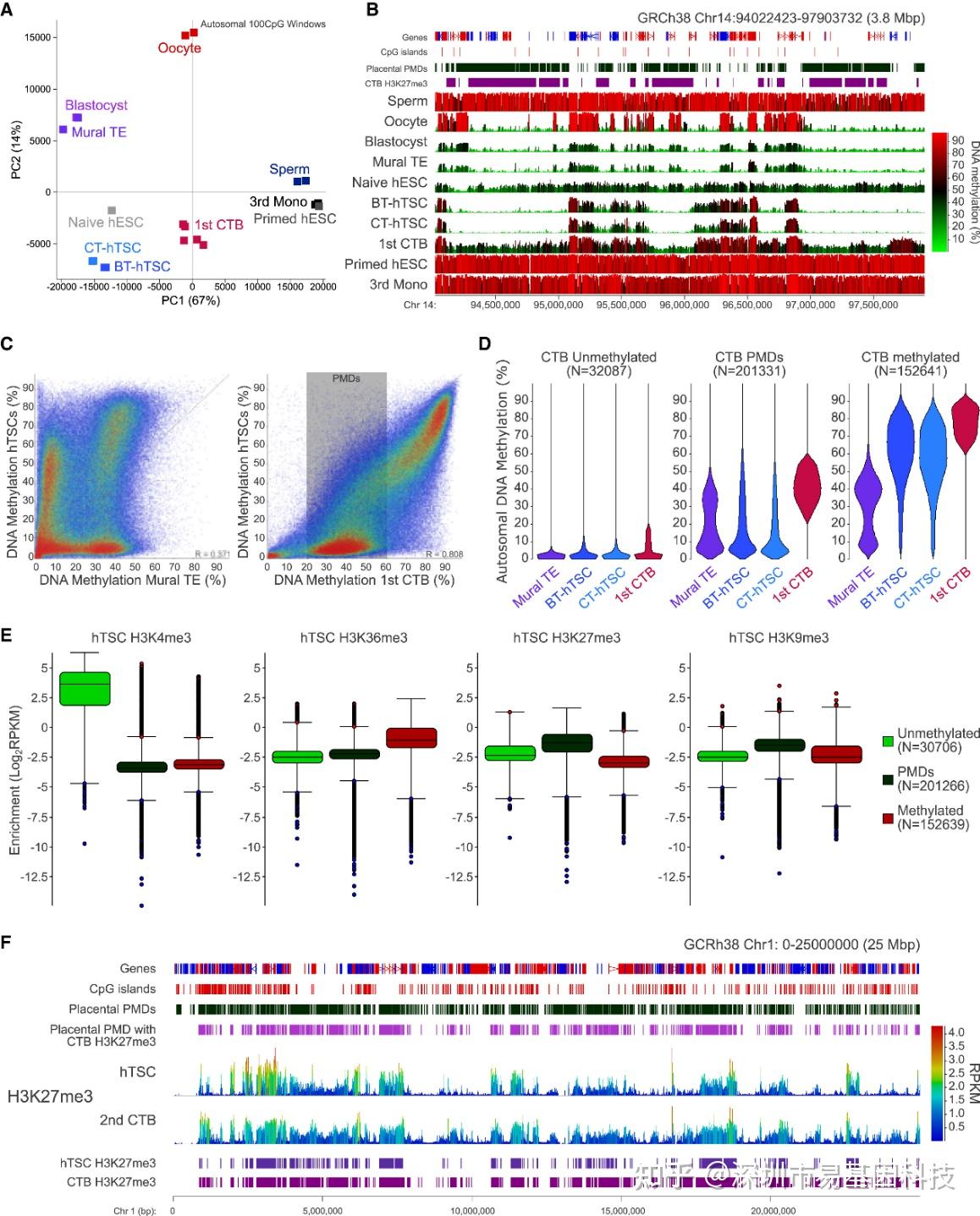
图1:与体内细胞类型相比,hTSCs表现出特异性DNA甲基化谱
(A)基于常染色体100-CpG窗口的DNA甲基化数据,生物学重复和技术重复样本聚集模式的主成分分析。hTSCs的甲基化谱与TE和1st CTB均不重叠,表明其甲基化图谱具有特异性。
(B)截图显示培养的naive和primed hESCs、来自囊胚的hTSCs(BT-hTSCs,n=1)以及来自第一孕期CTBs的hTSCs(CT-hTSCs,n=1)的DNA甲基化模式,与精子(n=3)、卵母细胞(N=2)、囊胚(n=1)、滋养层外细胞(mural TE,n=2)、第一孕期CTBs(n=5)和第三孕期胎儿单核细胞(3rd Mono,n=3)的生物学重复样本的平均DNA甲基化水平进行比较。
(C)左侧散点图显示了滋养层外细胞(mural TE,n=2)与hTSCs(n=2)的常染色体100-CpG窗口的平均DNA甲基化水平,右侧散点图显示了第一孕期CTBs(n=5)与hTSCs(n=2)的比较。
(D)豆点图显示TE(n=2)、BT-hTSCs(n=1)和CT-hTSCs(n=1)的100-CpG窗口的DNA甲基化水平,与第一孕期CTBs(n=5)进行比较,覆盖范围从未甲基化区域(<20%)到部分甲基化区域(20%-60%)再到高甲基化区域(>60%)。
(E)箱线图显示了hTSCs中H3K4me3(n=5)、H3K36me3(n=2)、H3K27me3(n=3)和H3K9me3(n=3)的组蛋白修饰平均富集水平:H3K4me3(活性标记)富集于未甲基化区域;H3K36me3(转录相关)与基因体甲基化共定位;H3K27me3和H3K9me3(抑制性标记)在hTSCs的PMDs中富集度低于体内CTBs。
(F)截图显示了hTSCs(n=3)和第二孕期CTBs(2nd CTB,n=2)的H3K27me3富集(RPKM)水平。图中还标注胎盘PMDs和在CTBs中也富集H3K27me3的PMDs的子集,以及hTSCs和CTBs中的H3K27me3富集区域。
(2)培养条件对hTSCs的DNA甲基化模式影响有限
研究发现,不同的培养条件(如氧气浓度、培养基类型、3D培养等)对hTSCs的DNA甲基化模式影响有限。结果显示,尽管在某些条件下(如低氧环境)DNA甲基化水平有轻微变化,但这些变化不足以恢复胎盘PMDs(Figure 2)。这表明hTSCs的DNA甲基化模式相对稳定,且不易通过常规培养条件进行调控。
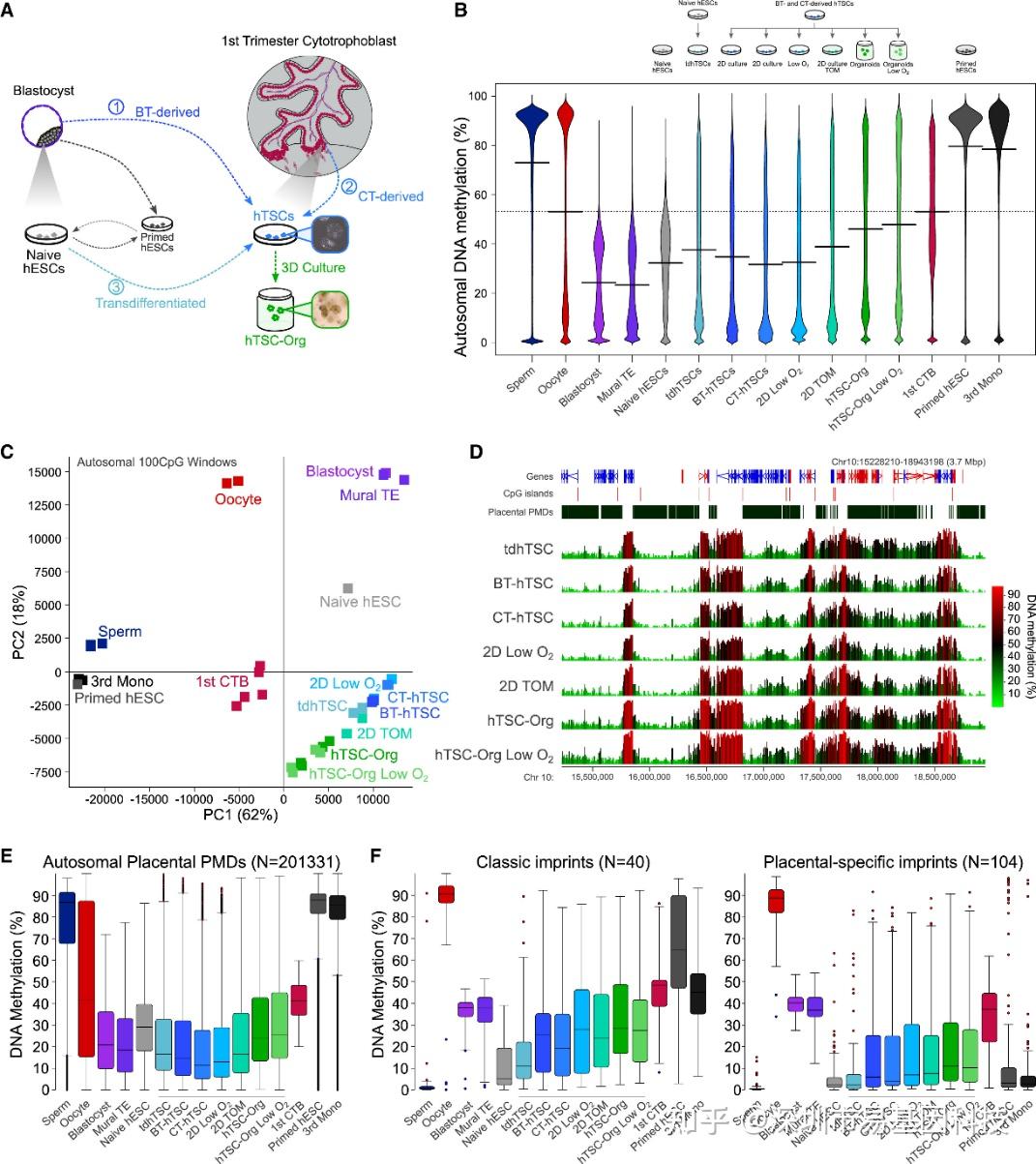
图2:不同细胞来源、培养条件和氧浓度均未能恢复胎盘甲基化表征
- 示意图展示hTSCs的不同来源,包括囊胚来源①、细胞滋养层细胞来源②以及从naive hESCs转分化而来③,以及本研究中使用的培养方法。
- 豆点图显示人类卵细胞(n=2)、精子(n=3)、囊胚(n=1)、滋养层外细胞(n=2)、naive hESCs(n=1)、转分化hTSCs(tdhTSCs,n=2)、BT-hTSCs(n=1)、CT-hTSCs(n=1)、低氧条件下的2D培养hTSCs(2D low O2,n=2)、滋养层类器官培养基中的2D培养hTSCs(2D TOM,n=2)、作为类器官培养的hTSCs(hTSC-Org,n=2)以及低氧条件下的hTSC-Orgs(n=2)、第一孕期CTBs(n=5)、primed hESCs(n=1)和第三孕期胎儿单核细胞(3rd Mono,n=3)的常染色体100-CpG窗口平均DNA甲基化水平。
- 主成分分析显示常染色体100-CpG窗口的DNA甲基化,不同技术重复和生物学重复呈现聚类模式。(D) 截图显示每种hTSC条件下的100-CpG窗口的DNA甲基化水平,覆盖范围为10号染色体上的3.7 Mbp区域。图中还标注第一孕期CTBs中定义的胎盘PMDs。
(E) 箱线图显示各样本中胎盘PMDs的100-CpG窗口的平均DNA甲基化水平。
(F) 箱线图显示所有样本中经典印记DMRs(左,不同组织中印记的DMRs)和胎盘特异性印记DMRs(右)的平均DNA甲基化水平。
(3)DNA甲基转移酶(DNMT)活性改变可能支持hTSCs中非典型DNA甲基化
研究发现,hTSCs中DNA甲基转移酶(DNMTs)的表达模式与原代滋养层细胞不同。hTSCs中DNMT3B表达较高,而DNMT3A和DNMT3L表达较低。DNMT3B主要在转录基因体内发挥作用,这与hTSCs中DNA甲基化的基因体定位一致。通过单细胞RNA测序分析,研究者发现hTSCs的DNMT表达模式与早期胚胎发育中的滋养层细胞相似,但DNMT3L表达缺失。这表明DNMT3L缺失可能是hTSCs中DNA甲基化模式异常的主要原因。
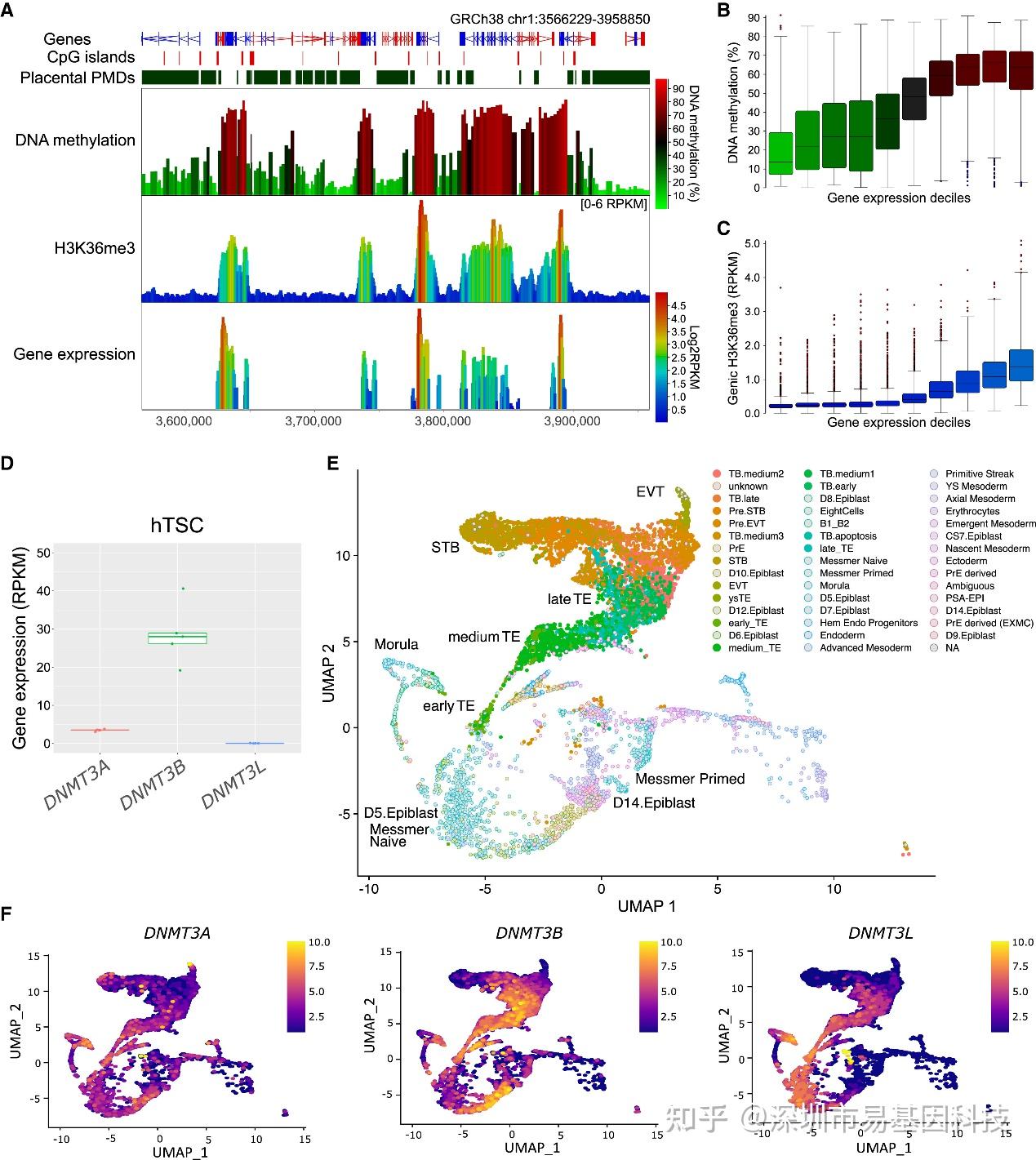
图3:hTSCs中的DNA甲基化富集在具有H3K36me3标记的转录基因体中,并与DNMT3B表达相关。
- hTSCs中平均DNA甲基化(n=2)、H3K36me3富集(n=2)和基因表达(n=5)截图。
- 箱线图显示hTSCs中基因表达水平的十等分位的基因DNA甲基化水平。
- 箱线图显示hTSCs中基因表达水平的十等分位的基因H3K36me3水平。
- 箱线图显示hTSCs(n=5)中DNMT3A、3B和3L的mRNA表达水平。
- UMAP图显示来自人类胚胎和干细胞的单细胞RNA-seq数据,按细胞类型着色。
- UMAP图(E)上显示了NMT3A、3B和3L的表达水平。
(4)在hTSCs中异位表达DNMT3L可恢复胎盘PMDs
通过慢病毒介导的DNMT3L过表达实验,研究者发现DNMT3L的异位表达能够显著恢复hTSCs中的胎盘PMDs。与对照组相比,DNMT3L过表达的hTSCs显示出更高的DNA甲基化水平,特别是在PMDs区域。此外,DNMT3L过表达还恢复了H3K27me3标记的CpG岛和印记区域的中间甲基化水平。这表明DNMT3L在胎盘PMDs的建立中起着关键作用。
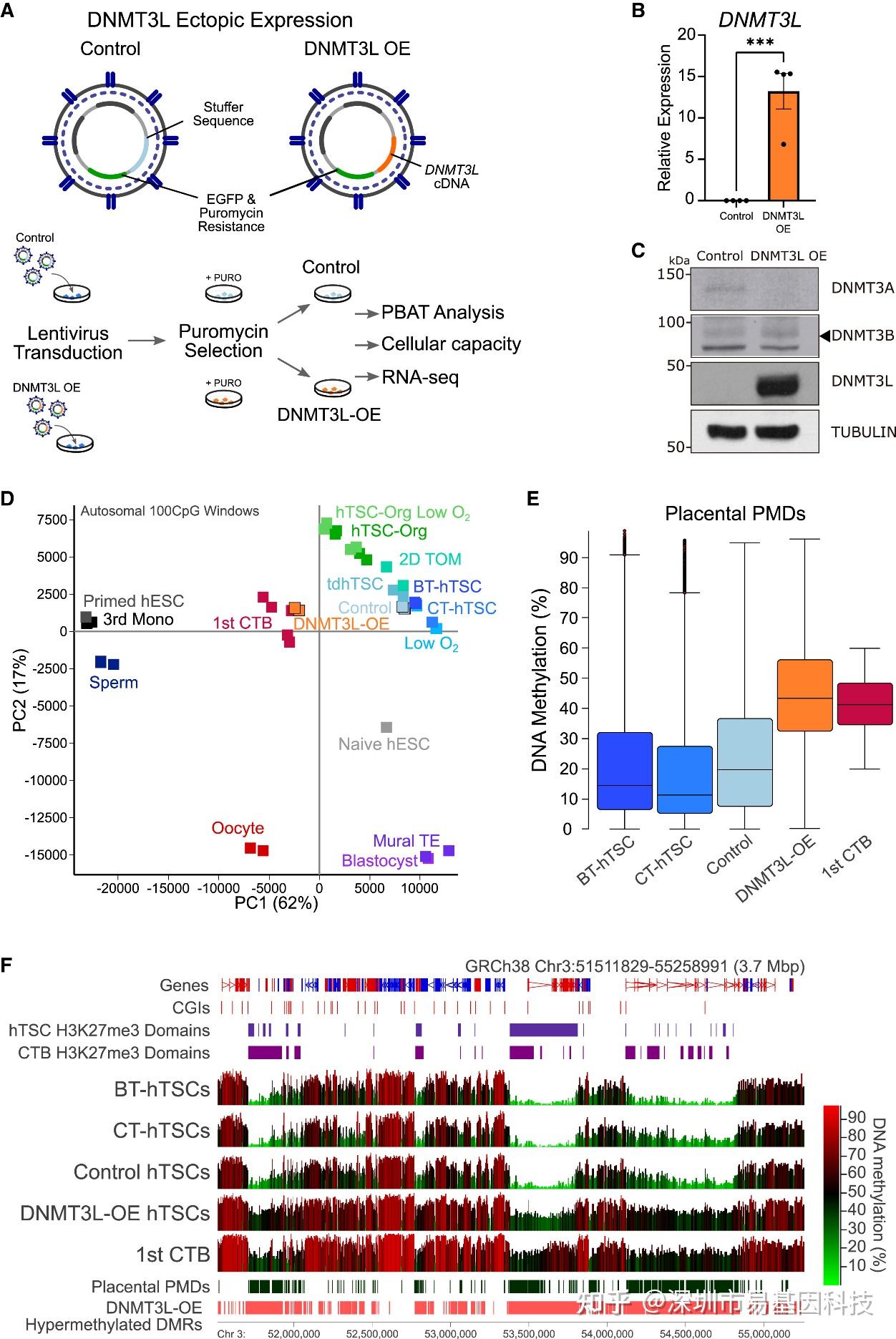
图4:DNMT3L异位表达可恢复胎盘PMD
- hTSCs中DNMT3L慢病毒表达系统示意图,展示载体设计(上)和用于获得稳定表达DNMT3L的hTSCs以及转导对照细胞(下)的实验流程。
- 对照(Stuffer)细胞和DNMT3L过表达(DNMT3L-OE)hTSCs的DNMT3L表达RT-qPCR分析。
- Western blot分析显示对照组与DNMT3L-OE hTSCs中的DNMT3A、DNMT3B、DNMT3L和TUBULIN,证实DNMT3L在DNMT3L-OE hTSCs中的表达。
- 主成分分析显示基于常染色体100-CpG窗口的DNA甲基化,揭示DNMT3L-OE hTSCs与第一孕期CTBs最为接近。
- BT-hTSCs(n=1)、CT-hTSCs(n=1)、对照hTSCs(n=3实验重复)和DNMT3L-OE hTSCs(n=3实验重复)中胎盘PMDs内100-CpG窗口与第一孕期CTBs(n=5)比较的平均DNA甲基化水平箱线图。
- T-hTSCs、CT-hTSCs、对照hTSCs、DNMT3L-OE hTSCs和第一孕期CTBs中3号染色体3.7 Mbp区域的100-CpG窗口的平均DNA甲基化水平截图,特别是在PMDs内DNA甲基化水平增加。图中还标注在第二孕期CTBs或hTSCs中定义的H3K27me3区域、在第一孕期CTBs中定义的胎盘PMDs以及与对照组相比DNMT3L-OE中高甲基化的差异甲基化区域(DMRs)。
5. DNMT3L过表达的hTSCs具有相似自我更新能力,但在类器官培养中分化能力受损
DNMT3L过表达的hTSCs在2D培养中显示出与对照组相似的自我更新能力,但在3D类器官培养中,其分化为合体滋养层细胞(STBs)的能力受到抑制。研究发现,DNMT3L过表达的hTSCs在类器官培养中表现出CREB蛋白水平降低和CREB激活减少,这可能与CRE序列基序的超甲基化、CREB蛋白水平降低以及STB分化相关的信号通路受损有关。这一结果表明,DNMT3L的持续表达可能影响hTSCs的分化能力,特别是在STBs的形成过程中。
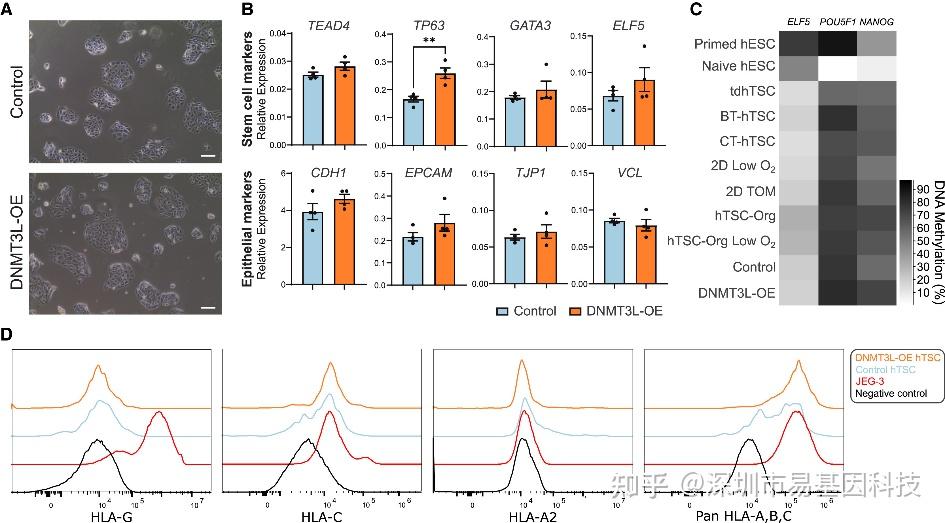
图5:DNMT3L过表达(DNMT3L-OE)hTSCs在自我更新和细胞身份上与对照hTSCs相似
- 图像显示DNMT3L过表达(DNMT3L-OE)与对照hTSCs相似的细胞形态。
- 条形图显示对照hTSCs和DNMT3L-OE中干细胞标记物TEAD4、TP63、GATA3和ELF5(上)以及上皮细胞标记物CDH1、EPCAM、TJP1和VCL(下)的相对表达水平RT-qPCR。
- 热图显示hESC基因OCT4(POU5F1)和NANOG以及hTSC基因ELF5的转录起始位点100-CpG窗口的DNA甲基化。展示primed hESCs(n=1)、naive hESCs(n=1)、tdhTSCs(n=2)、BT-hTSCs(n=1)、CT-hTSCs(n=1)、低氧条件下2D培养的hTSCs(n=2)、TOM(2D TOM)中的hTSCs(n=2)、hTSC-Org(n=2)以及低氧条件下的hTSC-Orgs(n=2)、对照hTSCs(n=3实验重复)和DNMT3L-OE hTSCs(n=3实验重复)的平均值。
- 直方图展示DNMT3L-OE和对照hTSCs、JEG3细胞(阳性对照)以及阴性对照中HLA-G、HLA-C、HLA-A2和泛HLA-A,B,C的表达情况。
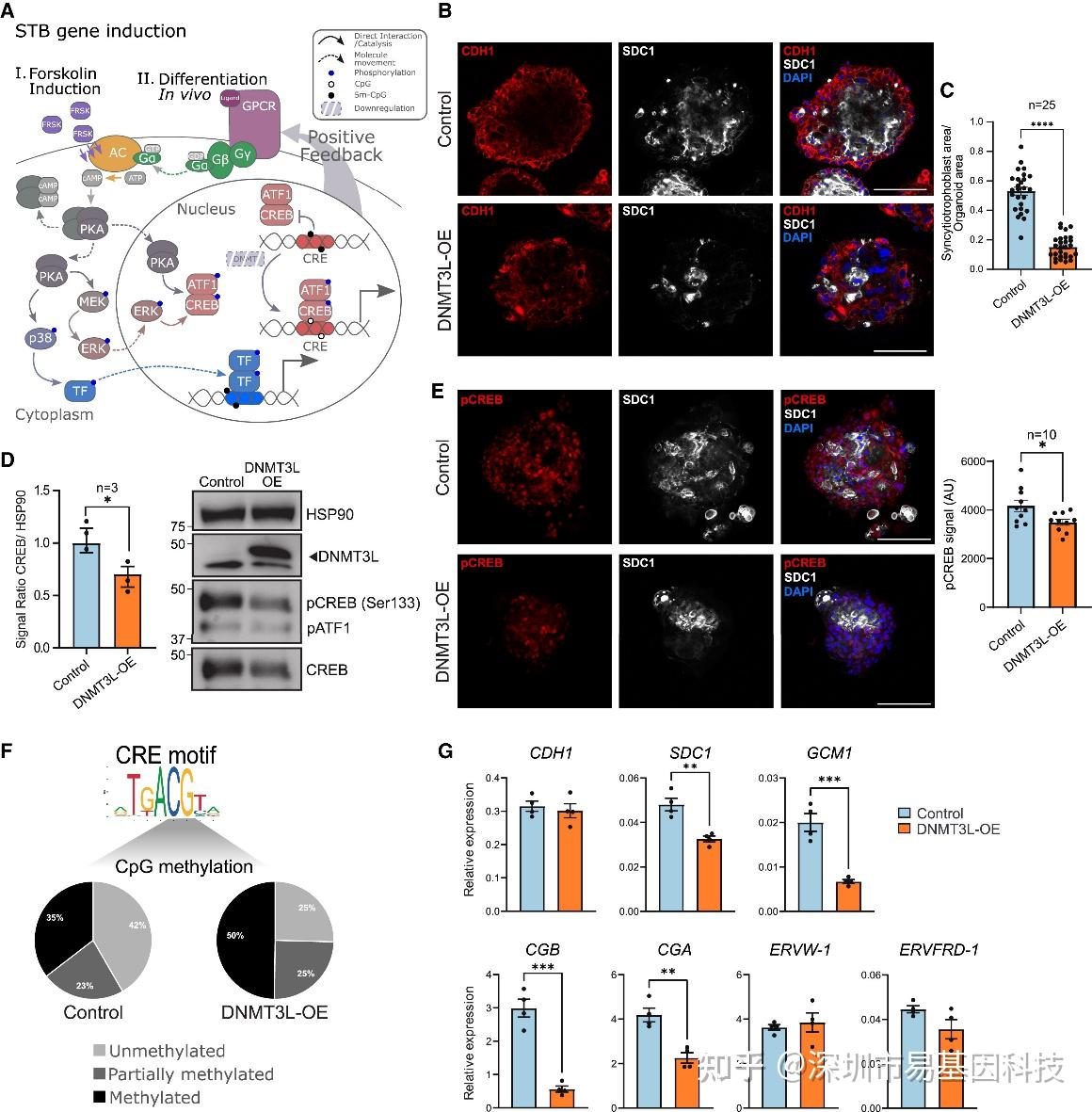
图6:在类器官培养中,DNMT3L过表达hTSCs无法有效分化为合体滋养层细胞
- 示意图展示在hTSCs中诱导合体滋养层细胞(STB)形成的信号级联反应。具体而言,STB分化可以通过2D培养中的FRSK(I)或类器官培养来模拟体内分化(II),这通过GPCR激活实现,据推测包括hCG与其受体LHCGR的结合。腺苷酸环化酶(AC)的活通过将ATP转化为cAMP来诱导STB分化,激活蛋白激酶A(PKA),进而激活MAPK级联反应,最终通过磷酸化激活CREB和ATF-1转录因子(TF),并结合到cAMP反应元件(CREs,红色DNA基序)。CRE motif的DNA甲基化会阻断CREB:ATF-1的结合,而DNMT表达的缺失被认为会导致CRE motif的非甲基化。由MAPK激活的对甲基化不敏感转录因子(蓝色)可以结合到目标motif。
- 免疫荧光图像显示与对照hTSC类器官相比,DNMT3L-OE hTSC类器官中的CDH1和SDC1。
- 条形图显示DNMT3L-OE和对照hTSC类器官中STB面积相对于总类器官面积的比例。
- western blot显示与对照组相比,DNMT3L-OE hTSC类器官中的HSP90、DNMT3L、pCREB(Ser133)、pATF-1和总CREB丰度。左侧图表显示CREB条带强度的定量。与对照组相比,DNMT3L-OE hTSC类器官显示出CREB蛋白水平的降低。
- 免疫荧光图像显示了对照和DNMT3L-OE hTSC类器官中的pCREB和SDC1。
- 显示了被ATF-1结合的经典CRE motif,其中包含一个中心CpG位点。
- 条形图显示通过RT-qPCR检测DNMT3L-OE和对照hTSC类器官中上皮细胞标记物CDH1和STB标记物SDC1、GCM1、CGB、CGA、ERVW-1(SYNCYTIN-1)以及ERVFRD-1(SYNCYTIN-2)的相对表达。
结论和启示
本研究揭示了hTSCs的DNA甲基化模式与原代滋养层细胞的显著差异,并通过DNMT3L的异位表达恢复了胎盘PMDs。研究结果表明,DNMT3L在胎盘PMDs的建立中起着关键作用,而其在hTSCs中的缺失可能是导致DNA甲基化模式异常的主要原因。此外,DNMT3L过表达的hTSCs在类器官培养中表现出分化能力受损,这可能与CREB结合位点的DNA甲基化增加有关。这些发现为理解胎盘发育的表观遗传调控提供了重要信息,并为研究胎盘相关疾病提供了新的模型和方法。
微量WGBS技术的作用
微量WGBS技术在本研究中发挥了关键作用。它允许研究者对少量细胞样本进行全基因组DNA甲基化分析,揭示了hTSCs与原代滋养层细胞之间的DNA甲基化差异。这一技术的应用不仅提高了研究的分辨率,还为研究胎盘发育的表观遗传调控提供了新的工具。未来,类似的研究可以利用微量WGBS技术,深入探索其他细胞类型和组织的DNA甲基化模式,揭示其在发育和疾病中的作用。
目前,易基因科技有限公司在DNA甲基化修饰等表观遗传检测领域,积累了一系列国际先进技术,可对高通量单细胞甲基化组学研究,微量DNA甲基化测序,ctDNA、FFPE等严重降解痕量DNA甲基化检测等,提供多种有效解决方案。低成本、高通量的单细胞甲基化组学测序技术建立,将为研究者对不同时间和空间的单细胞表观修饰研究奠定基础。
- 微量细胞或单细胞全基因组甲基化测序(Micro DNA-WGBS)DNA起始量:
单细胞/100-1000个细胞
1ng基因组DNA
90%以上基因组CG覆盖 - 微量细胞或DNA简化基因组甲基化测序(Micro DNA-RRBS)DNA起始量:
1ng基因组DNA;
10-20M有效CG位点覆盖;
10-20G测序数据量; - 微量cfDNA简化基因组甲基化测序(cfDNA-RBS)
100ul血浆或1ng cfDNA
10M有效CG位点覆盖
15-20G测序数据量
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化、cfDNA)和表观转录组学(m6A、m5C、m1A、m7G、ac4C、RNA与蛋白互作)、DNA与蛋白互作及染色质开放性技术方案(ChIP-seq、ATAC-seq),更多表观组学或多组学研究可关注易基因公众号、网站、市场微信号等,期待与各位老师开展合作交流,详询易基因:0755-28317900。
参考文献:
Lea G, Doria-Borrell P, Ferrero-Micó A, Varma A, Simon C, Anderson H, Biggins L, De Clercq K, Andrews S, Niakan KK, Gahurova L, McGovern N, Pérez-García V, Hanna CW. Ectopic expression of DNMT3L in human trophoblast stem cells restores features of the placental methylome. Cell Stem Cell. 2024 Dec 31. doi: 10.1016/j.stem.2024.12.007.
相关阅读:
1. Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
2. Nature | 易基因DNA甲基化测序助力人多能干细胞向胚胎全能8细胞的人工诱导
3. 项目文章 | WGBS+RNA-seq揭示黄瓜作物的“源-库”关系受DNA甲基化调控
4. 项目文章 | WGBS等揭示SOX30甲基化在非梗阻性无精症中的表观遗传调控机制
5. 项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
6. 项目文章 | WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
7. 项目文章:微量DNA甲基化分析揭示MeCP2在卵子发生和卵巢衰老中的表观遗传调控
8. 项目文章 | 单细胞DNA甲基化与转录组分析揭示猪生发泡卵母细胞成熟的关键调控机制
9. 项目文章 | 植入前胚胎的全基因组DNA甲基化和转录组分析揭示水牛胚胎基因组激活进展
10. 项目文章:微量DNA甲基化分析揭示MeCP2在卵子发生和卵巢衰老中的表观遗传调控
11. 项目文章|微量WGBS+ACE-seq揭示卵巢早衰的人卵丘细胞DNA甲基化与羟甲基化表观基因组图谱
12. 项目文章:微量DNA甲基化分析揭示MeCP2在卵子发生和卵巢衰老中的表观遗传调控
13.项目文章|DNA甲基化与转录组联合分析助力揭示花青素生物合成调控柑橘果实品质的表观新机制
DNA 5mC专题
