分享一篇由阿三团队近期发表在Plant Commun上的文章:PTF-Vāc: An explainable and generative deep co-learning encoders-decoders system) for ab-initio discovery of plant transcription factor binding sites。该研究开发了一个名为 PTF-Vāc 的新型人工智能系统,用于植物中转录因子结合位点(TFBS)的从头发现(ab-initio discovery)。

PTF-Vāc(梵语“Vāc”意为“言说”,寓意“道出”结合位点)系统基于一个通用的“转录因子-DNA互作”模型,通过深度协同学习转录因子的三维结构变异和其结合位点的序列变异,成功地将TFBS的发现过程与预定义的基序模型完全解耦。PTF-Vāc不仅能够从头、精准地预测任意物种中任意转录因子的结合位点,甚至能区分同一基因不同剪接变体的结合偏好,其性能在全面的基准测试中远超现有先进算法。
背景与意义
- • 背景
- • 植物中转录因子(TF)与DNA结合位点(TFBS)具有高度的物种间变异性和上下文依赖性。
- • 现有工具大多依赖于预定义的TF特异性模型或实验数据(如ChIP-seq),难以应对跨物种、跨条件的TFBS预测。
- • 植物基因组复杂、重复序列多,TFBS的识别更具挑战性。
- • 研究意义
- • 提出一个通用、可解释、生成式深度学习系统PTF-Vāc,摆脱对实验数据和TF特异性模型的依赖。
- • 可用于新物种基因组注释、剪接变体分析、全基因组TFBS预测等,推动植物调控研究。
方法概述
- • 核心模型
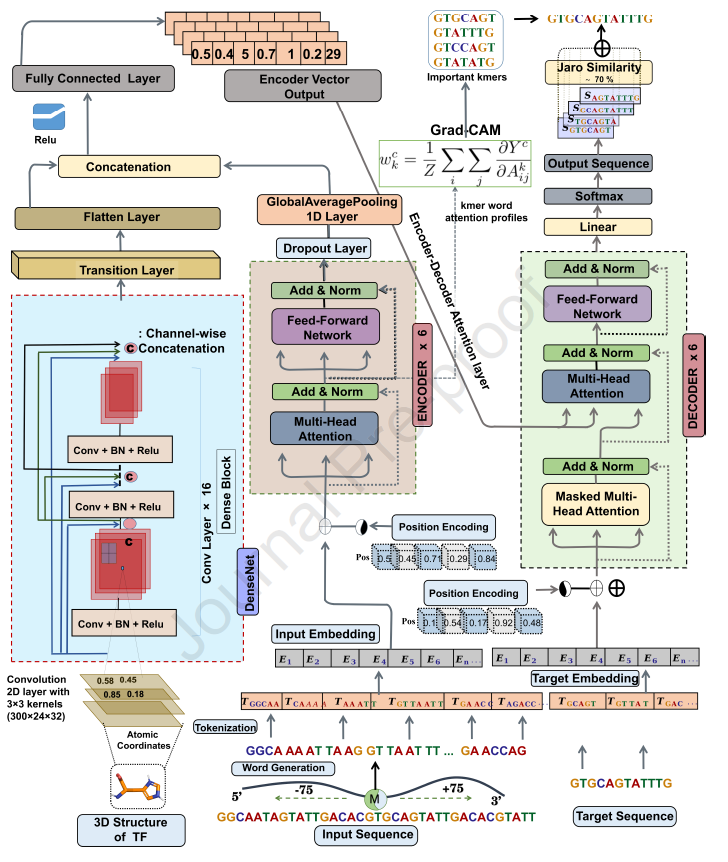
- • PTF-Vāc 基于前期开发的 PTFSpot(一个TF结构与其结合区域协同学习的通用模型)。
- • 使用Transformer编码器-解码器结构结合DenseNet处理TF的3D结构信息,实现序列到序列的生成式学习。
- • 输入:DNA序列(150–162 bp)+ TF的AlphaFold2预测结构。
- • 输出:精确的TFBS序列(9–12 bp)及其motif。
- • 训练数据
- • 使用拟南芥(Arabidopsis thaliana)中40个TF家族的48,000个结合区域。
- • 采用五聚体至七聚体(pentamer–heptamer)词汇表示DNA序列,提升模型表达能力。
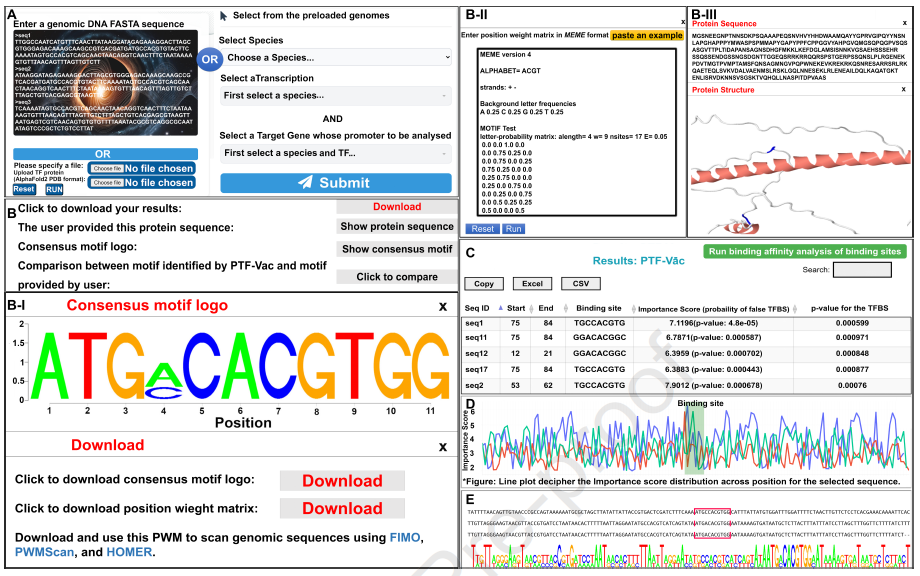
- • 可解释性
- • 引入 Grad-CAM 对模型进行可视化解释,识别影响TFBS预测的关键序列片段和结构区域。
主要结果
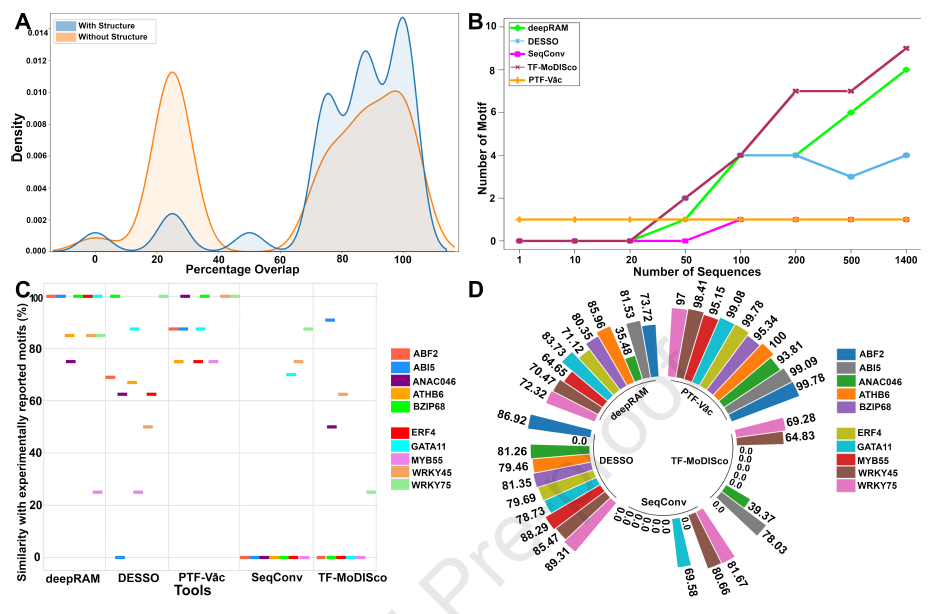
1. 模型性能
- • 在测试集上准确率达到 92.76%,优化后提升至 93.2%。
- • 去除TF结构信息后准确率下降至 73.9%,证明结构信息对预测至关重要。
2. 实验验证
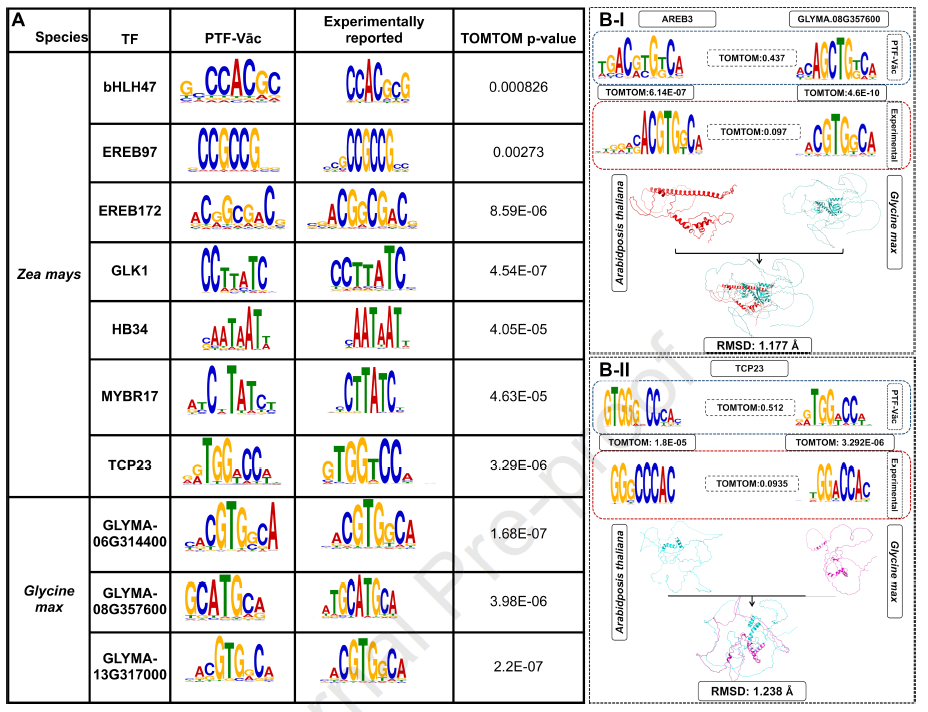
- • 与JASPAR数据库中实验验证的motif进行比对:
- • 所有36个TF的预测motif均显著匹配实验结果(TOMTOM p < 0.01)。
- • 其中13个TF的motif匹配度达 100%。
- • 结合位点覆盖实验数据的比例为 83.26%–100%。
3. 分子对接验证
- • 对5个TF进行分子对接分析,PTF-Vāc预测的TFBS与实验位点的结合能几乎一致,甚至更优,验证其生物学合理性。
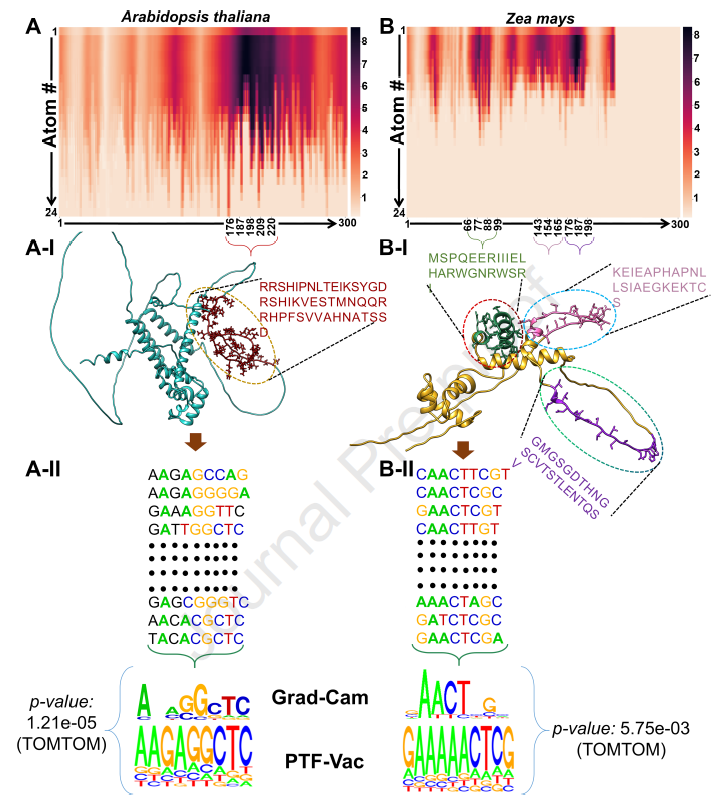
4. 跨物种应用
- • 在玉米(Zea mays)和大豆(Glycine max)中测试,PTF-Vāc仍能准确识别TFBS,表现出良好的跨物种泛化能力。
5. 剪接变体分析
- • 成功识别拟南芥ARF8两个剪接变体的不同结合位点,揭示其结构差异导致结合特异性变化。
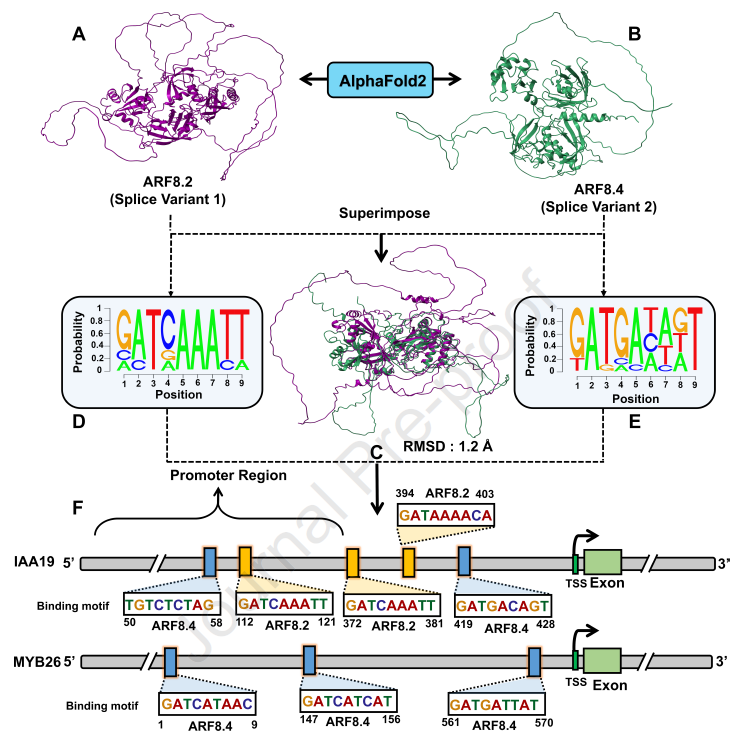
6. 全基因组应用
- • 在茶树(Camellia sinensis)全基因组中预测BES1转录因子的靶基因:
- • 预测1,878个靶基因,其中519个与ChIP-seq结果一致(一致性达98%)。
- • 功能富集分析显示其参与干旱、盐胁迫响应通路,符合已知生物学功能。
结论与展望
- • PTF-Vāc 是一个无需实验数据、无需预定义motif、可解释、跨物种通用的TFBS发现工具。
- • 它突破了传统方法对实验数据和TF特异性模型的依赖,适用于:
- • 新物种基因组注释
- • 剪接变体调控差异研究
- • 全基因组TFBS预测
- • 提供了在线服务器(https://scbb.ihbt.res.in/PTF-Vac/)和**开源代码**(https://gitlab.com/scbblab/ptfvac),便于科研社区使用与扩展。
- • 当前仅适用于植物,但方法可拓展至动物等其他生物领域。
局限性与未来方向
- • 需要TF的3D结构(目前依赖AlphaFold2预测)。
- • 当前仅针对植物TF进行训练和验证。
- • 尚不支持条件特异性结合(如组织、发育阶段、胁迫响应等)预测。
