研究背景
全基因组选择(genomic selection, GS)是现代分子育种中的一项重要技术,作为一种强大的机器学习GS方法,堆叠集成学习(stacking ensemble learning, SEL)有效地融合了不同模型(基学习器)的优势,以精确描绘表型与基因型之间的复杂关系。然而,在SEL的关键步骤中,目前缺乏一种有效且统一的基学习器选择框架,而且仅根据经验选择固定的基学习器并不总是合理的。所以,有必要开发一种能够根据数据本身的特征从大量候选模型中自适应选择高性能基学习器,从而构建高效SEL模型,并对模型可解释性进行探索。
论文概要
2025年8月7日,鲁东大学麦类分子育种创新团队在《Theoretical and Applied Genetics》发表了题为“AdaptiveGS: an explainable genomic selection framework based on adaptive stacking ensemble machine learning”的研究论文。该研究开发了一种自适应且可解释的数据驱动型基学习器选择策略——adaptiveGS,用于为堆叠GS框架预筛选最优的基学习器,提高小麦产量性状基因组估计育种值(Genomic estimated breeding value,GEBV)预测准确性。进一步引入SHAP (the SHapley Additive explanations)方法对adaptiveGS的结果进行解释,可识别与小麦产量性状高度相关的显著单核苷酸多态性(SNPs),即主效应,并阐释SNPs间的交互效应。该研究不仅丰富了小麦产量性状全基因组选择算法,也有望在粮食增产、育种芯片设计及可持续农业发展等方面发挥作用。

主要研究结果
1.PR指数基学习器选择策略有效提升堆叠集成机器学习预测能力
本文利用皮尔逊相关系数(PCC)和归一化均方根误差(NRMSE)构建PR指数,通过PR指数从7个(或自定义)机器学习模型中自动筛选出排名前3的模型作为基学习器(图2),取代以往研究中事先根据经验直接指定基学习器的方式,从而使得算法能够根据数据自身特征自适应筛选高性能基学习器,进而组建高效堆叠集成算法(图1),提升其预测能力。
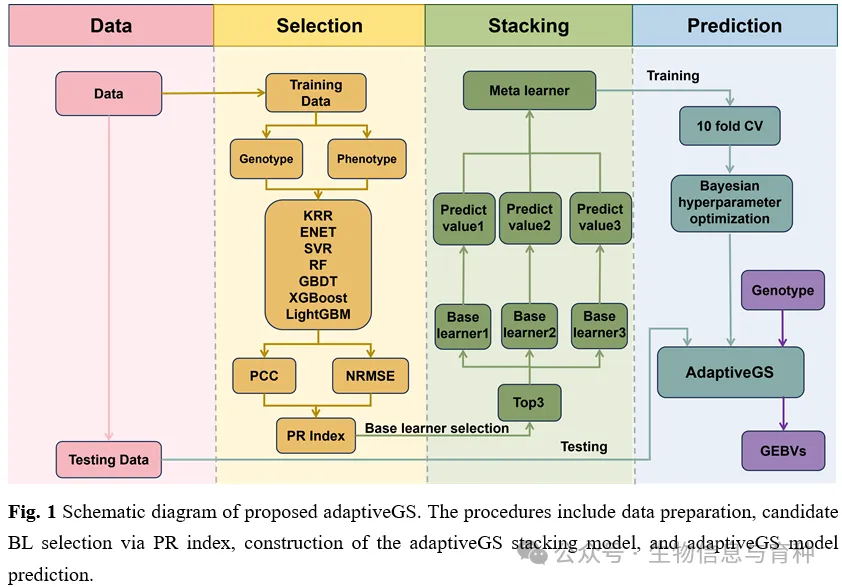
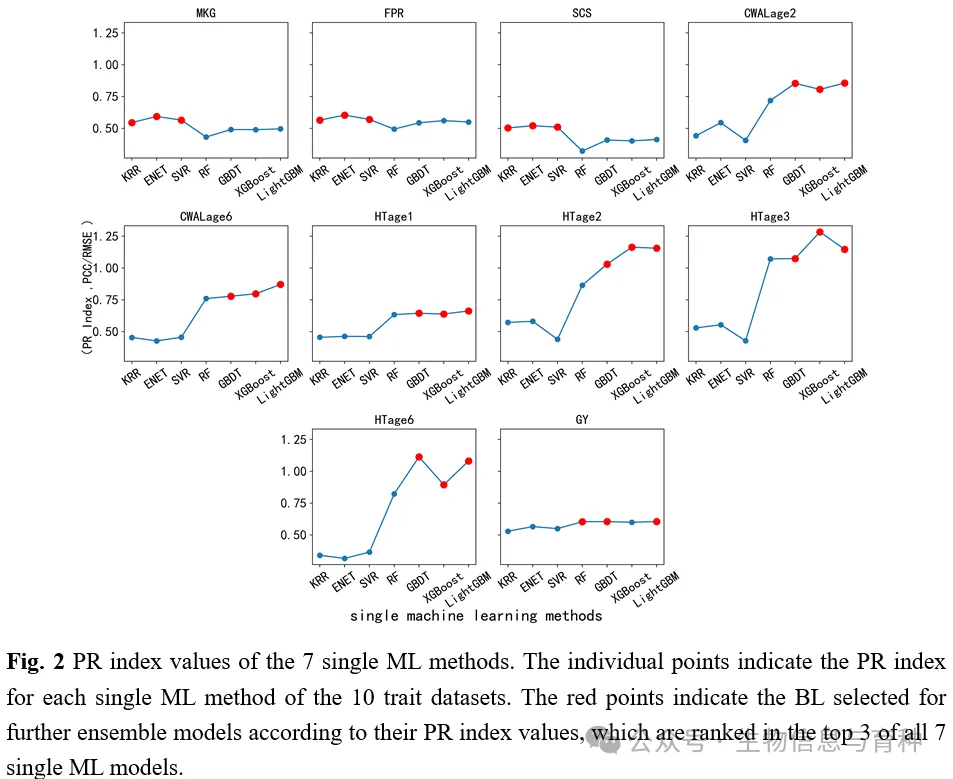
2.基于PR指数的adaptiveGS框架显著提升动植物育种GEBVs预测精度
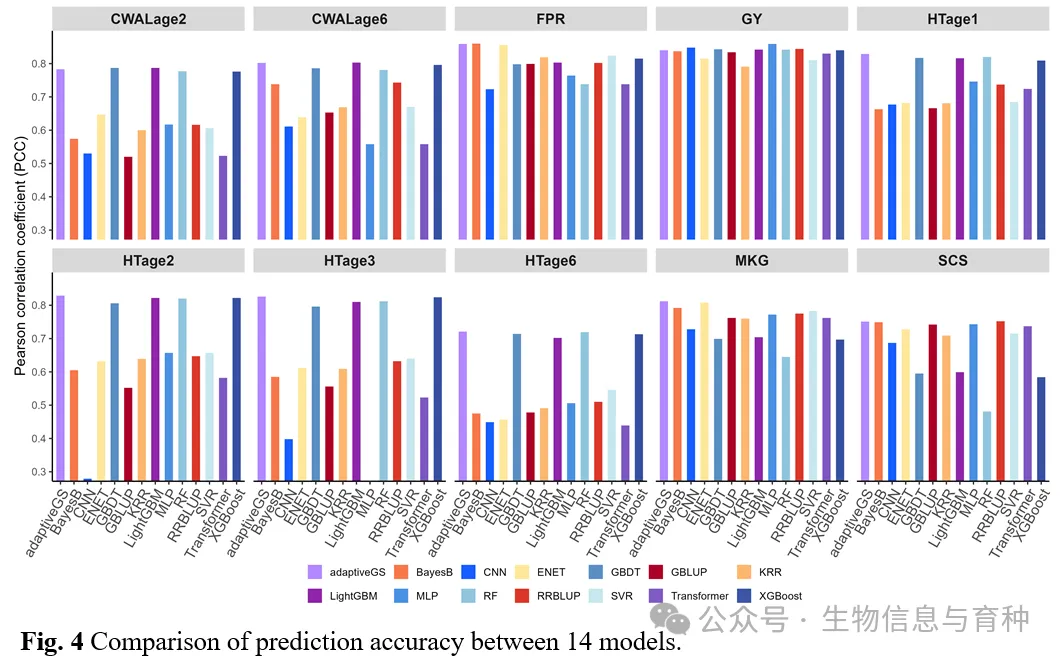
基于PR指数构建了adaptiveGS框架(图1),在包含小麦在内的4个动植物物种21个性状上,将adaptiveGS与其他13种GS算法进行了比较。结果表明,adaptiveGS在21个性状中的大多数上优于这13个模型,平均预测准确率(PCC)达到0.703,平均提高了14.4%(图4),展现出优异的预测准确性和稳健性。
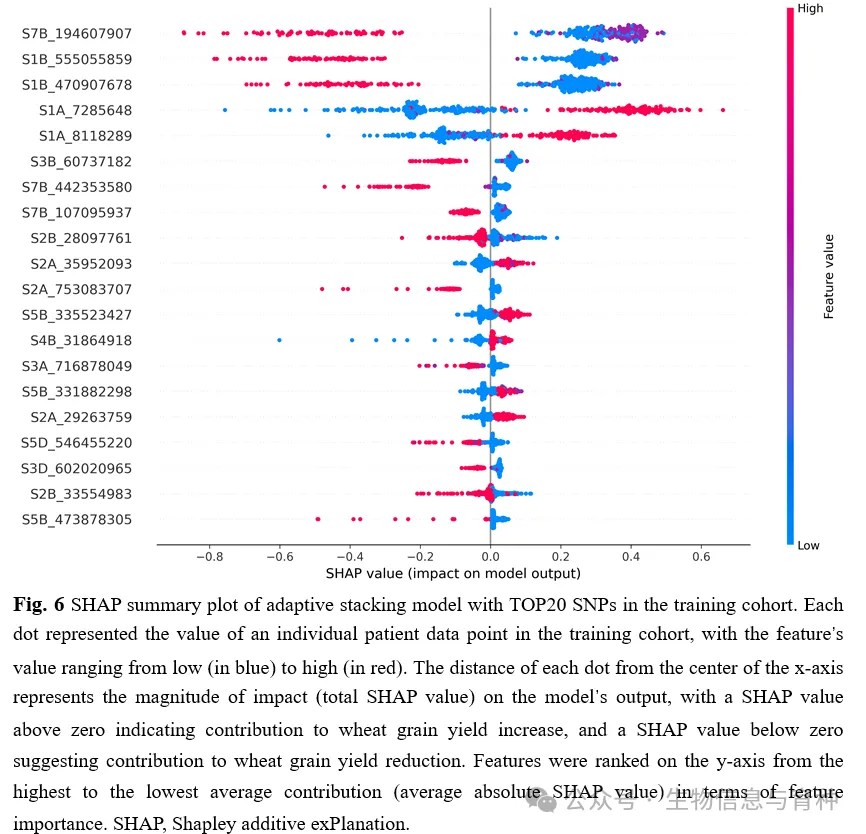
3.基于SHAP技术的adaptiveGS框架有助于解释小麦产量性状关键SNPs
利用SHAP事后解释技术对adaptiveGS进行解释,并识别影响小麦产量性状变异的显著SNPs以及SNPs之间潜在的相互作用效应。结果表明,本研究提出的adaptiveGS算法有助于识别可能影响小麦产量性状的潜在显著SNP位点,并可使用SHAP方法直观展示每个SNP对小麦产量性状GEBV预测的贡献(图6,图7,图8)以及SNPs之间的潜在相互作用效应(图9)。
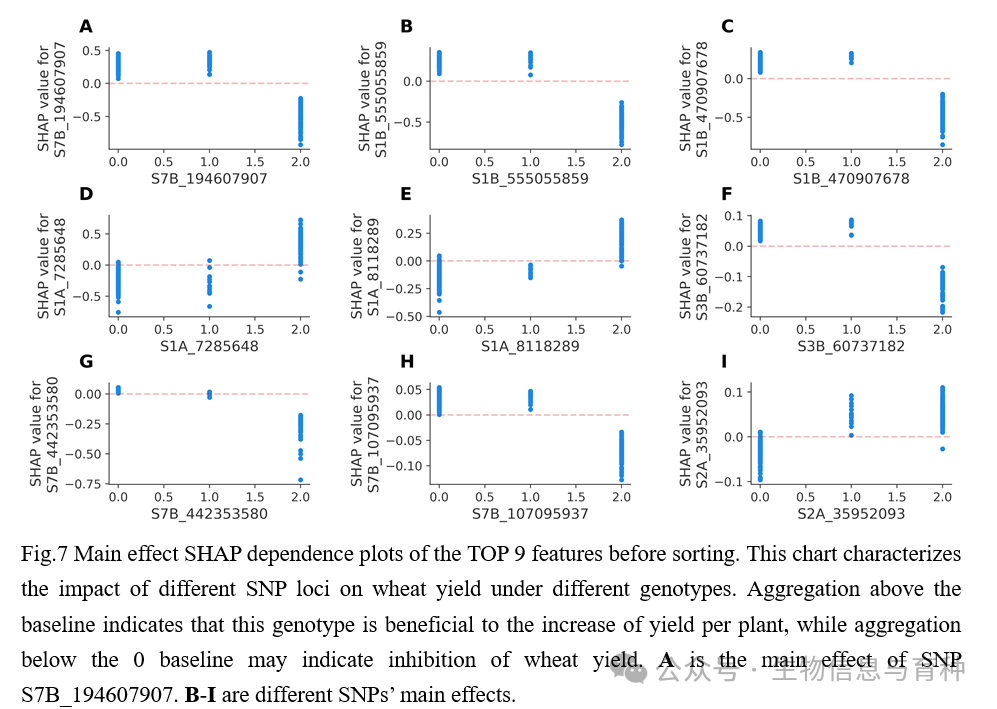
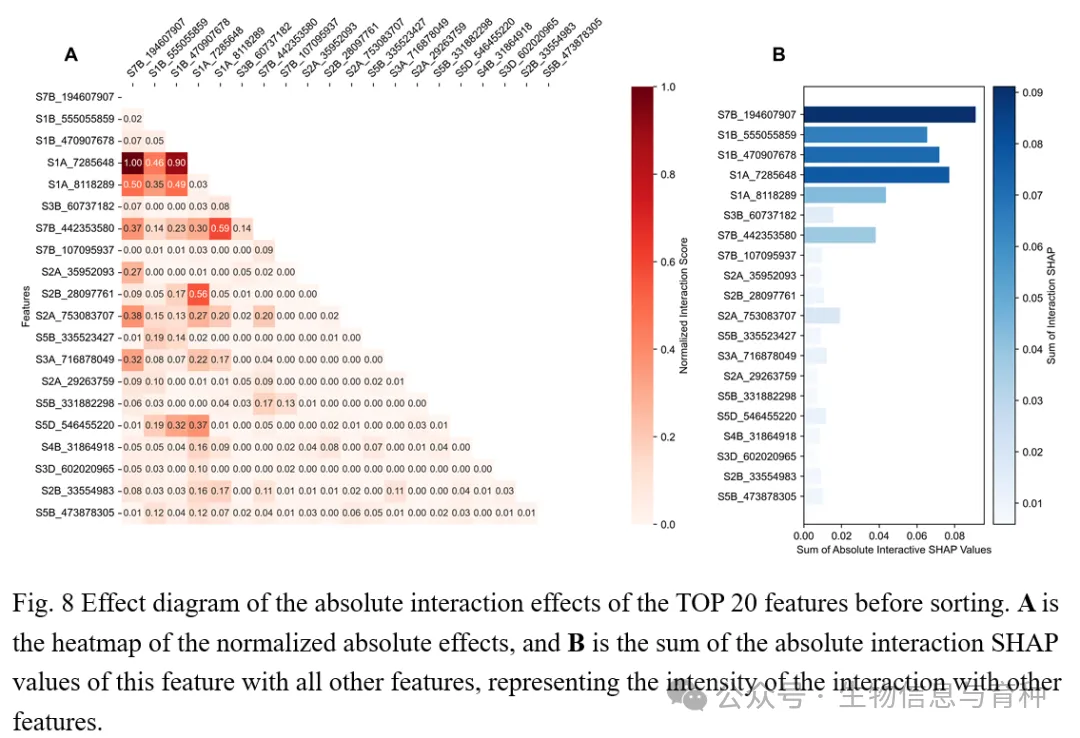
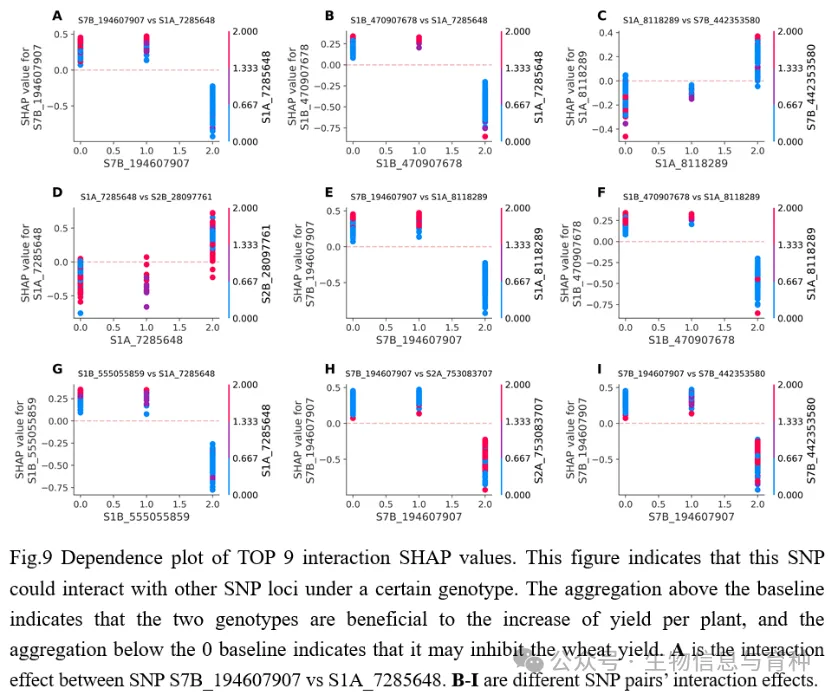
全文总结
在本研究中,我们开发了一种自适应且统一的堆叠集成机器学习全基因组选择框架,并设计了一种模型解释策略,以识别小麦产量性状的候选显著SNPs。针对基学习器选择中存在的不确定性问题,引入了基于皮尔逊相关系数(PCC)和归一化均方根误差(NRMSE)的PR指数,自动筛选最适配的基学习器构建堆叠基因组选择模型,该过程由输入的训练数据驱动,而非依赖用户经验,有助于为每一组表型和基因型数据“量身定制”精准的堆叠模型。将adaptiveGS与包括经典GS模型、机器学习和深度学习在内的13种模型的预测精度比较,发现adaptiveGS在大多数场景中优于所有13种模型。最后,通过SHAP值对adaptiveGS模型进行了解释,以提供GS研究中与特定性状相关的重要SNPs信息。本研究在有效提升GS模型小麦产量性状GEBV预测精度的同时,可较好地阐释影响小麦产量性状的若干关键SNPs,并为提升小麦产量、智能育种芯片设计以及推广到其他动植物育种领域等提供了理论依据和技术支撑。
