以cropformer为例:
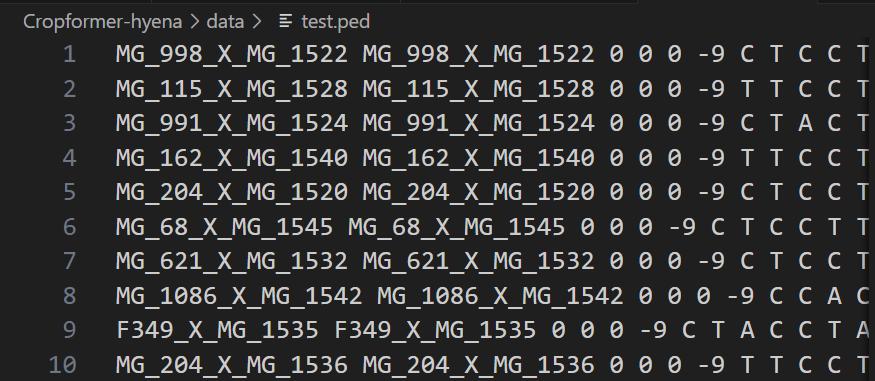
上图中每一行和每一列分别表示什么意思?
要理解 test.ped 文件中的数据含义,需结合作物基因组学中PED格式的标准定义和Cropformer的研究背景(基于基因组SNP数据预测作物表型),具体解析如下:
1. PED格式的通用结构
在作物/人类基因组研究中,PED文件是存储“样本系谱+基因型”的标准格式,每一行代表一个样本,列的含义为:
- 第1列:
Family ID(家系ID,若无复杂家系可与个体ID一致) - 第2列:
Individual ID(个体/样本唯一标识) - 第3列:
Paternal ID(父本ID,无则填0) - 第4列:
Maternal ID(母本ID,无则填0) - 第5列:
Sex(性别,作物研究中通常填0) - 第6列:
Phenotype(表型标签,此处填-9表示“未用于表型分类,仅作为基因型载体”) - 第7列及以后:SNP基因型(每个SNP位点的等位基因,按“A/T/C/G”等碱基对编码,每两个字符代表一个位点的基因型)
2. 结合图片的逐列解析
以图片中第1行数据 MG_998_X_MG_1522 MG_998_X_MG_1522 0 0 0 -9 C T C C T... 为例:
- 第1-2列:
MG_998_X_MG_1522
是样本的家系ID和个体ID,说明这是一个“自交系”或“杂交组合”样本(命名格式符合玉米/水稻育种材料的编号习惯)。 - 第3-4列:
0 0
表示该样本的父本、母本信息未知(或无系谱记录,作物研究中大量育种材料为纯系,此列常填0)。 - 第5列:
0
作物无性别区分,故填0。 - 第6列:
-9
表示该样本的表型未用于“分类任务”(Cropformer是“表型预测”任务,表型标签单独存储在test_label.csv中,此处PED仅承载基因型)。 - 第7列及以后:
C T C C T...
是SNP位点的基因型编码。每个SNP位点用两个字符表示(如C T代表该位点的两个等位基因),后续每两个字符对应一个SNP位点。这些碱基对是Cropformer模型的输入特征,用于学习“基因型→表型”的关联。
3. 在Cropformer中的角色
结合文献可知,Cropformer的核心是“从基因组SNP数据预测作物表型”。test.ped 是测试集的基因型文件,与 test_label.csv(测试集表型标签)一一对应:
test.ped提供“测试样本的SNP基因型”(模型输入);test_label.csv提供“测试样本的真实表型”(模型评估的对照标签)。
模型通过学习训练集的“基因型-表型”关联后,在test.ped的基因型上输出预测表型,再与test_label.csv的真实值对比,从而评估预测精度(如Pearson相关系数、均方误差)。
总结
test.ped 是Cropformer测试集中的“基因型数据载体”,遵循PED格式标准,存储了测试样本的系谱(简化为个体ID)和SNP基因型信息,是模型进行“基因组→表型”预测的核心输入数据之一。
